Delta-Delta Ct methode of Livak methode is de meest geprefereerde methode voor qPCR data analyse. Deze methode kan echter alleen worden gebruikt als aan bepaalde criteria is voldaan. Raadpleeg de aantekeningen bij het college om na te gaan of aan deze criteria is voldaan. Zo niet, dan is de meer algemene methode de Pfaffl methode. Lees het aanvullende leesmateriaal voor meer informatie over deze methode.
Hier volgen de stappen voor de Livak methode:
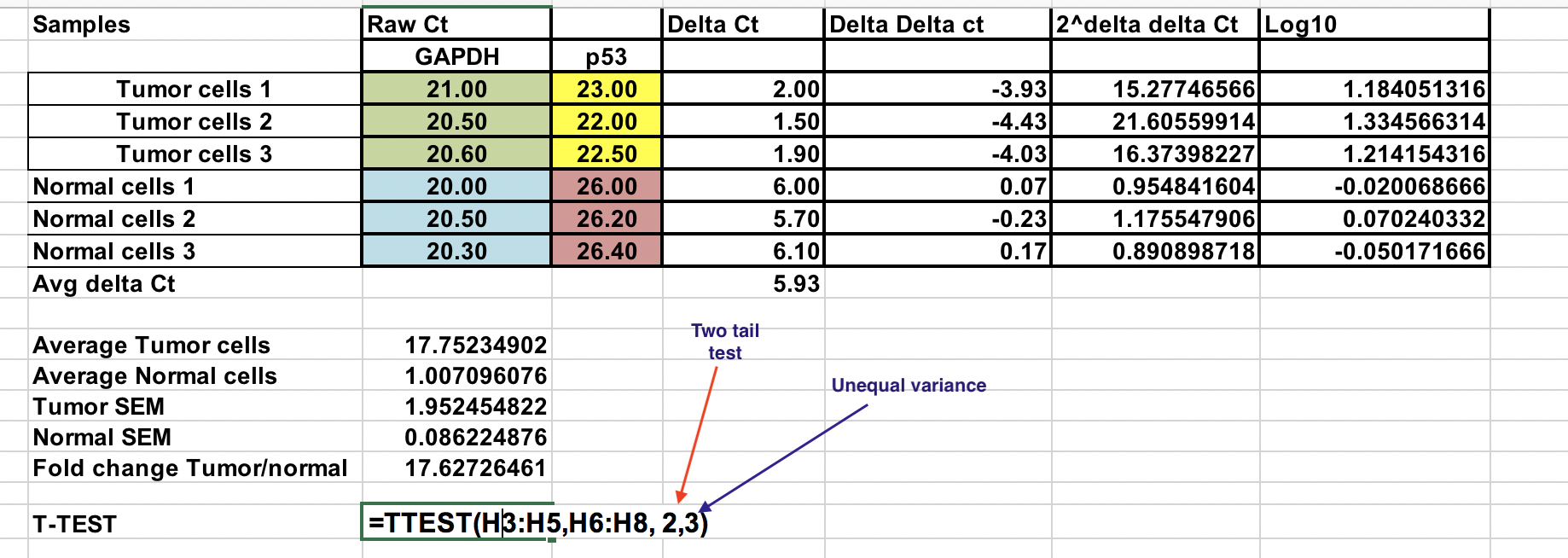
Het Excel bestand met alle berekeningen staan in de qPCR analyse map op Blackboard.
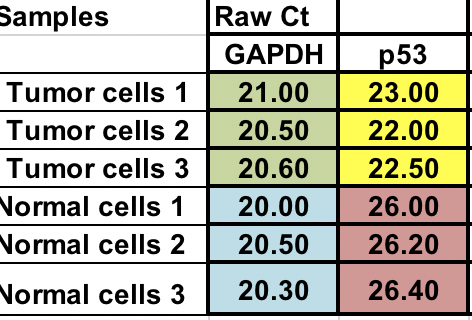
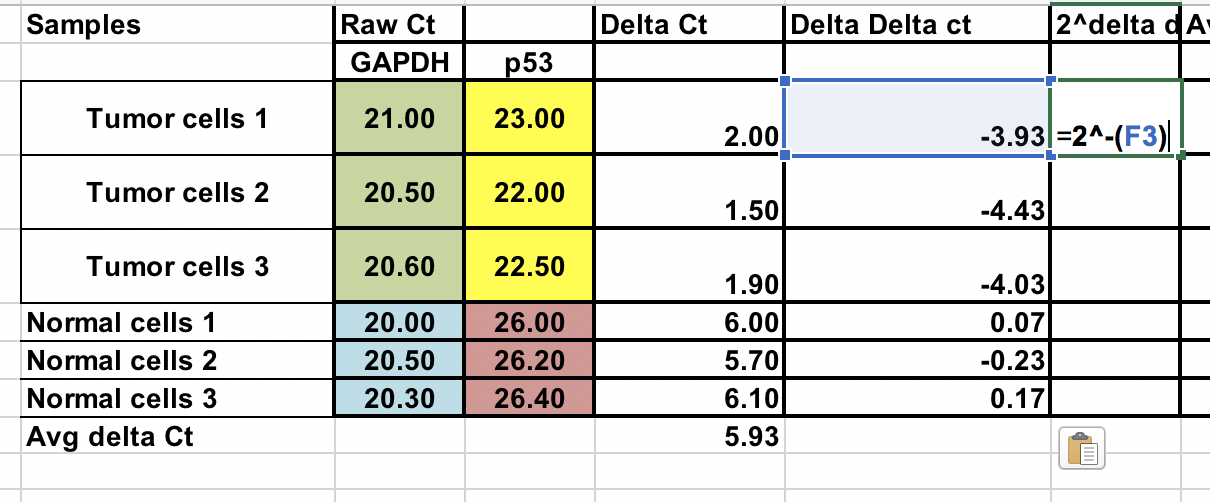
U hebt ruwe Ct (aantal cycli dat nodig is om de drempel te bereiken) voor normale en tumorcellen (3 replicaten voor elk).
Normalisatie¶
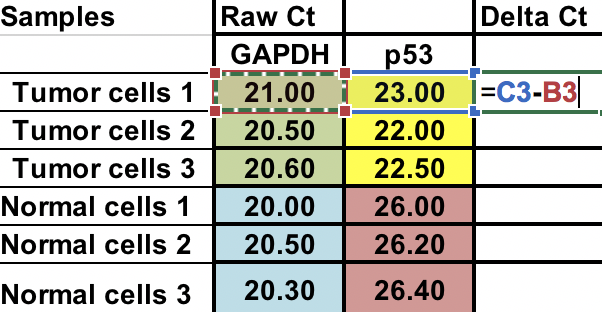
Eerst moet u het relatieve verschil berekenen tussen het gen van interesse (p53) en het huishoudgen (GAPDH).
∆Ct = Ct (gen van belang) – Ct (huishoudgen)
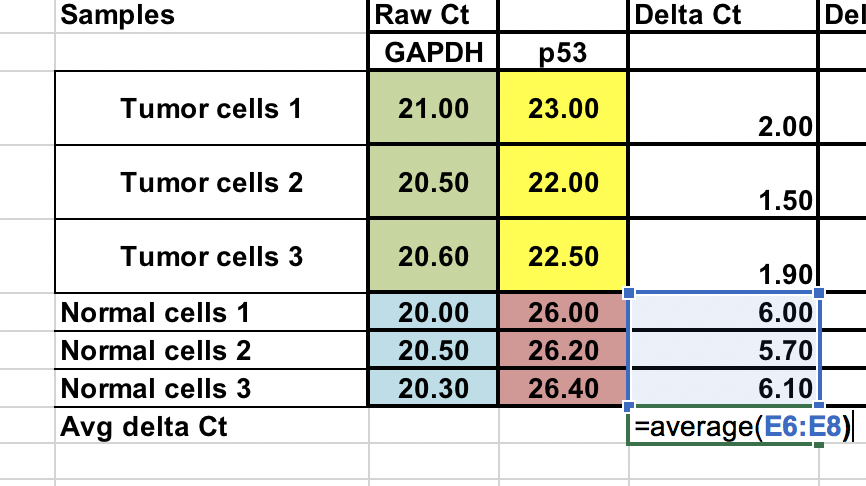
Gemiddelde van de controlemonsters (normale cellen)¶
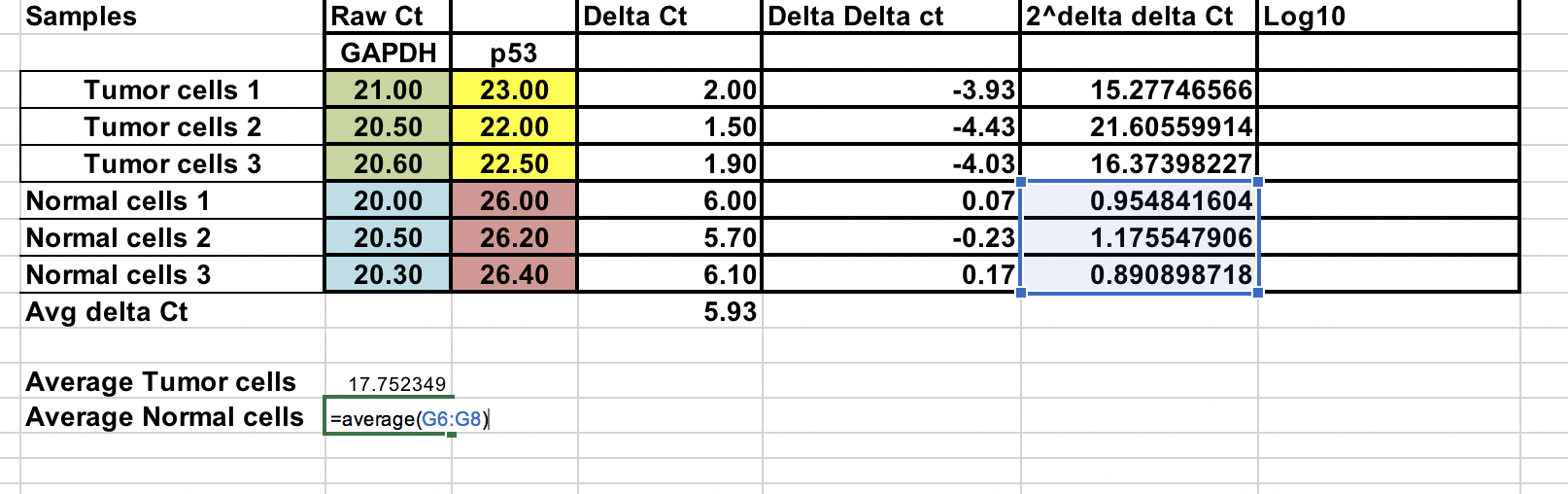
Als we onze tumor (behandeling) vergelijken met de controle (normale cellen), moeten we eerst het gemiddelde berekenen van de ∆Ct voor de 3 (normale) controlemonsters.
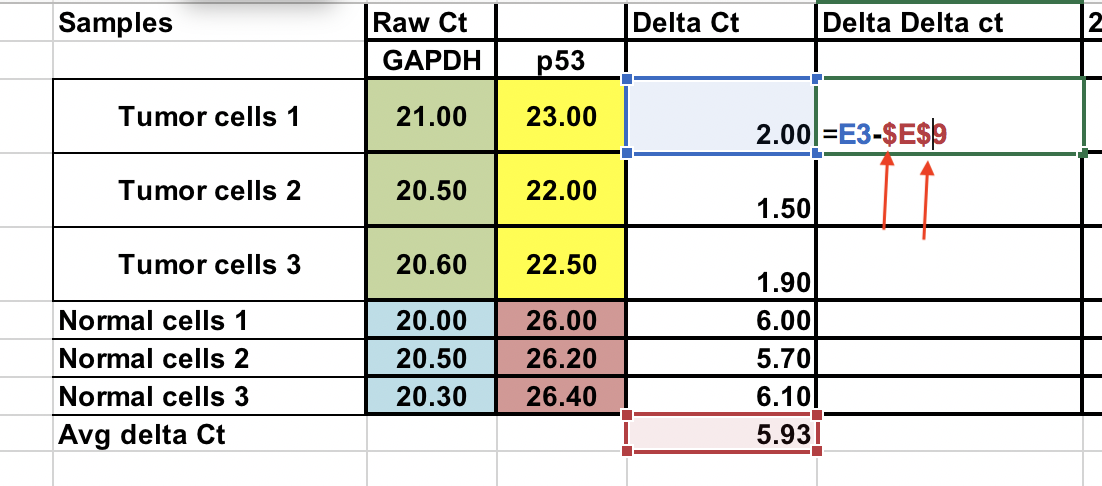
Bereken de ∆Ct ten opzichte van het gemiddelde van de ∆Ct normale cellen¶
∆Ct = ∆Ct (tumormonster) – ∆Ct (normaal gemiddelde)
U kunt dit ook met normale monsters doen. Gebruik $-tekens voor het kolomnummer en de ruwe letter (pijlen) om de cel vast te zetten.
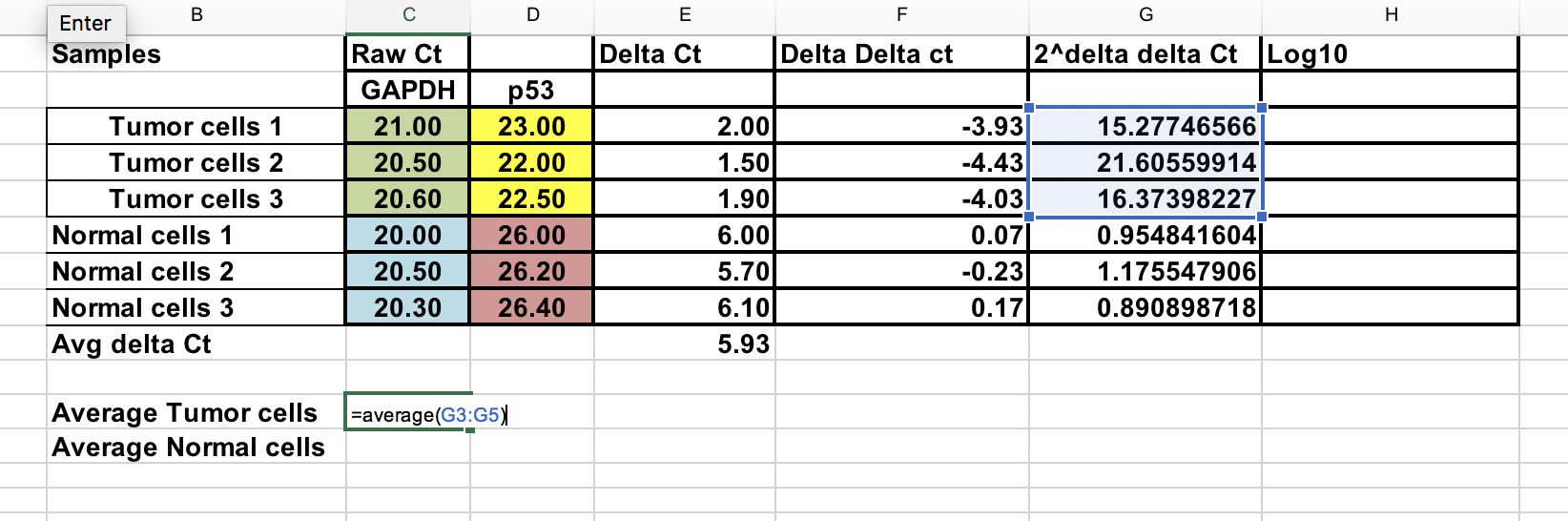
Voudige genexpressie voor elk monster¶
Zorg ervoor dat u de negatieve ∆∆Ct tot de macht twee verheft.
Voudige genexpressie = 2^-(∆Ct)
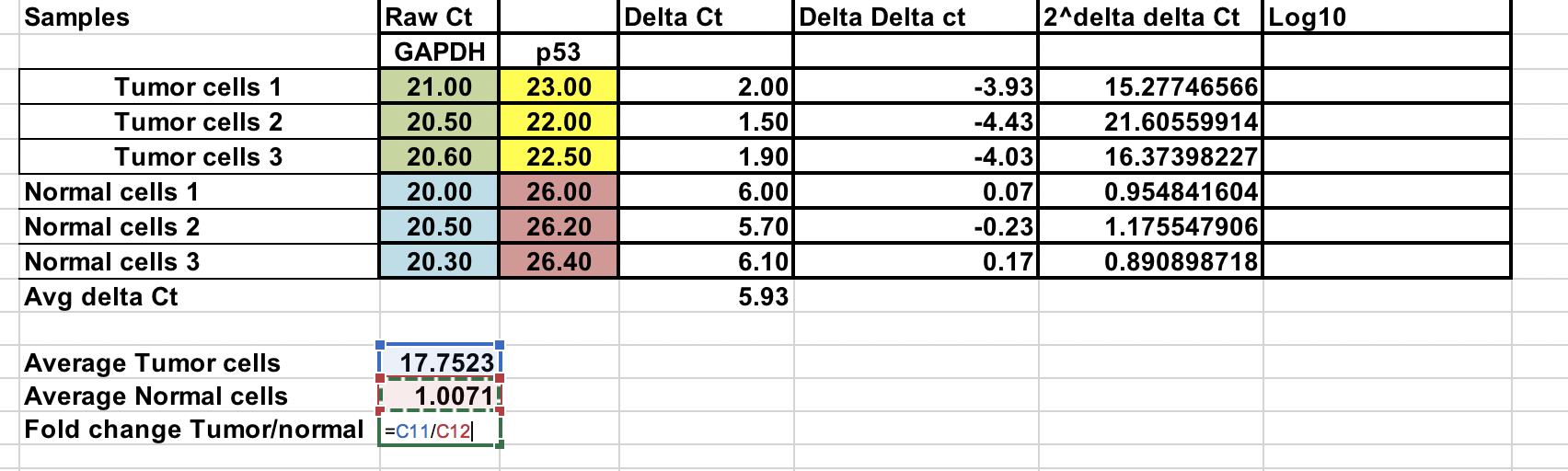
Overal voudige verandering¶
U kunt de gemiddelde voudige verandering berekenen voor zowel tumor- als normale monsters. De verhouding tussen deze twee is de vouwverandering tussen tumor- en normale monsters.
Log transformatie¶
Om parametrische statistische tests uit te voeren, zoals T-test, wordt geadviseerd de uiteindelijke genexpressieresultaten om te zetten in logwaarden (elke logbasis). Dit zou de verdeling van de gegevens symmetrisch maken.
Hier hebben we de 2^-(∆Ct) veranderd in log 10.
T-test¶
Voorzichtigheid is geboden bij het gebruik van parametrische tests indien de gegevens niet normaal verdeeld zijn, dit zou leiden tot foutieve conclusies.
Selecteer log 10 van 2^-(∆Ct) waarden voor Normale en tumormonsters zoals aangegeven. Gebruik twee-staarttest (nummer 2) en veronderstel ongelijke variantie (3).
De resulterende P-waarde is kleiner dan 0,05 en daarom verwerpen we de nulhypothese en twee steekproefmiddelen zijn significant verschillend op 0,05-niveau.